“统计大讲堂”第179讲回顾:利用图卷积网络整合分析空间转录组数据、空间位置和组织学图像数据
2021-12-23
12月15日下午,“统计大讲堂”系列讲座第179讲举行。本次讲座采取线上会议的方式,邀请南开大学统计与数据科学学院讲师李向杰作题为“利用图卷积网络整合分析空间转录组数据、空间位置和组织学图像数据”的报告。讲座由中国人民大学统计学院教授、应用统计科学研究中心研究员张景肖主持。

张景肖首先介绍了主讲人的相关信息。李向杰,南开大学统计与数据科学学院讲师,2019-2021 在中国医学科学院阜外医院从事博士后研究工作,2019博士毕业于中国人民大学统计学院,2017-2018获国家留学基金委公派资助在美国宾夕法尼亚大学交流学习,主要研究方向为统计基因组学、生物信息学,临床应用等相关研究。主持博士后面上基金一项,参与国自然重大项目一项。在Nature Methods, Nature Communications, Nature Machine Intelligence, Genome Research, cardiovascular research, Briefings in Bioinformatics, Journal of statistical planning of inference, Journal of Statistical Computation and Simulation,《统计研究》,《统计与信息论坛》等杂志发表20多篇论文。
李向杰首先对数据的背景进行介绍。随着科技发展,单细胞数据的发展也十分迅速。他首先介绍了生物学中三种研究方式——Bulk analysis、Single-cell analysis和Spatial analysis,并对三者进行比较。然后他讲述了空间转录组测序的优势,并且介绍了一些空间转录组数据库。
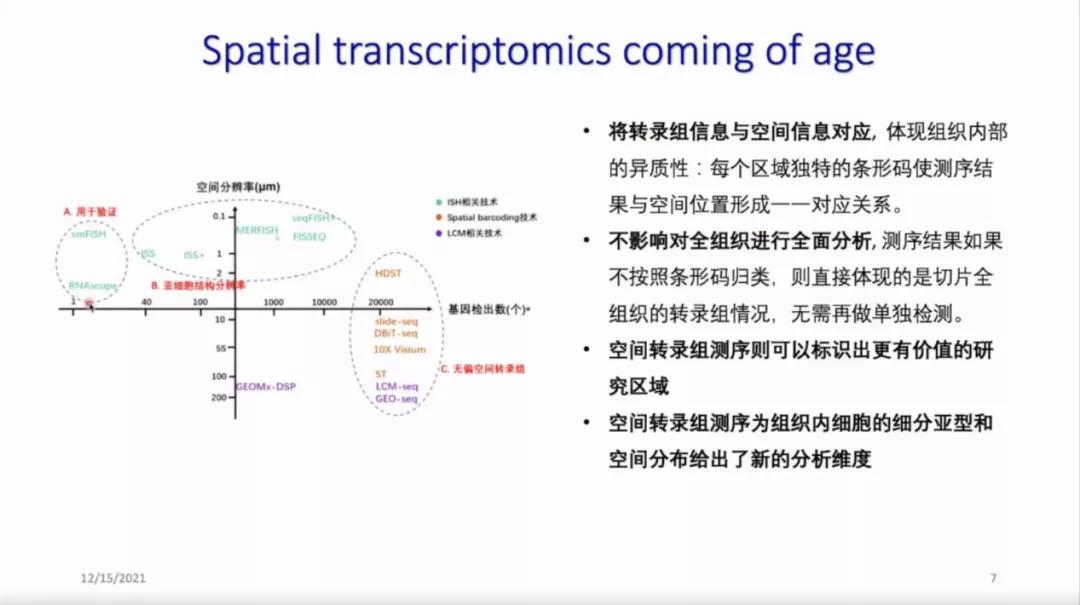
在简要介绍空间转录组的技术步骤后,他向我们展示了ST数据,并且分享了一些分析空间转录组数据的工具。
接下来,他介绍了图卷积神经网络。在处理组织信息时,邻接矩阵的构造需要空间转录组、组织学图像、各点物理位置的信息,然后聚类得到空间域。紧接着他进一步对SpaGCN方法的原理进行了说明。
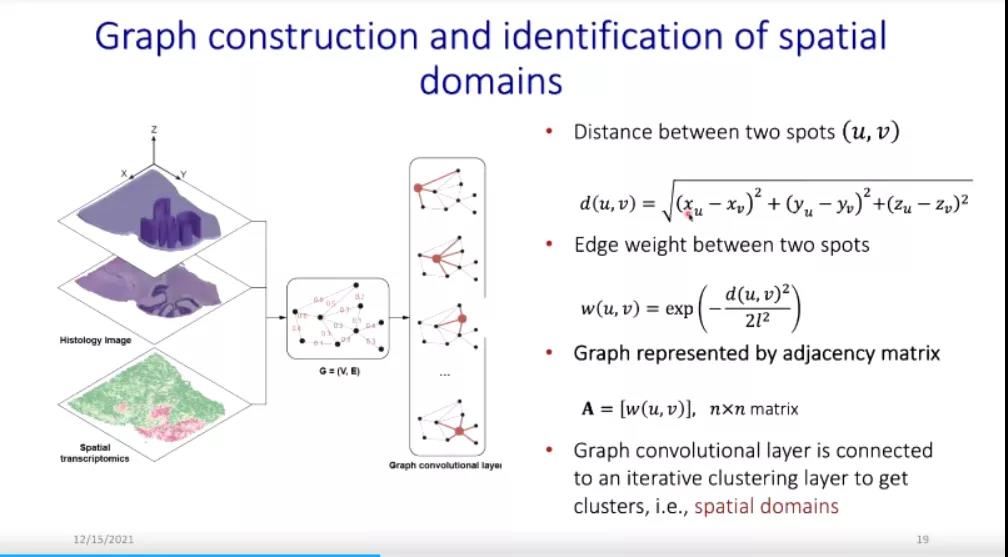
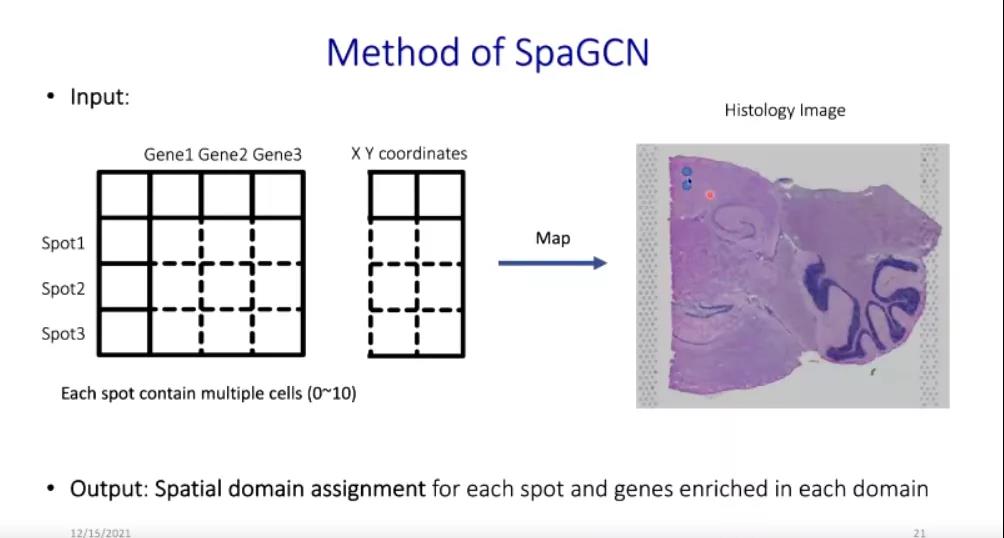
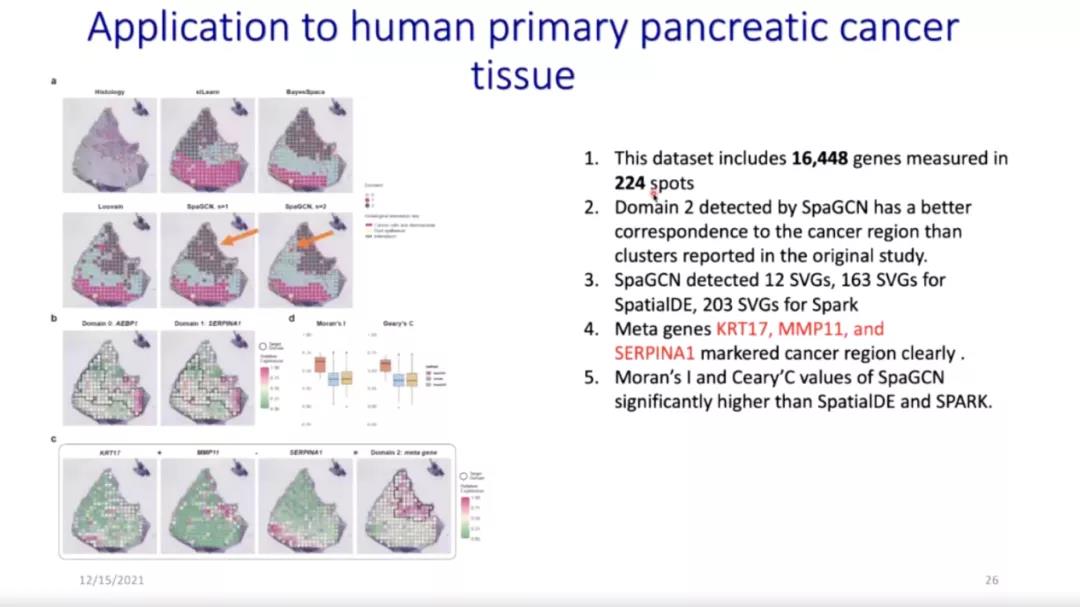
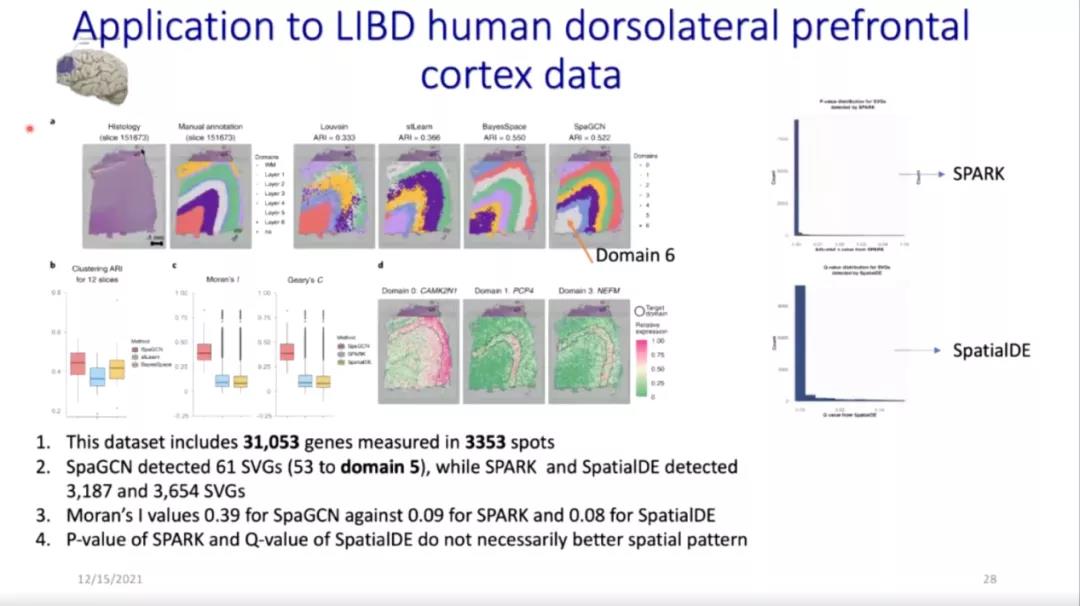
他给出一些数据集作为例子,对采用不同方法得到的结果进行比较,从而证明SpaGCN方法的优越性。第一个是关于乳腺癌的数据集,在小样本量的情况下通过SpaGCN方法得到了比较好的数据结果。第二个是LIBD人脑数据集,含有较多的切片数据。用Louvain、stLearn、BayesSpace、SpaGCN四种方法分别进行聚类得到图像后,SpaGCN在ARI、SVGs、Morgan’s I values的数值上均优于stLeam、BayesSpace方法。对于P值较小的基因,Spark和SpatialDE虽然能够探测更多SVGs,但与SpaGCN相比,二者在图像上不能与空间域进行较好的对应,数据代表性更低。第三个是小鼠后脑切片的数据集,在这一例子中对相似或相连的切片构造了新的邻接矩阵。第四个数据集是STARmap,在没有可用的图片数据时,引入HMRF和四种方法进行比较。第四个是关于小鼠嗅球的数据集,Spark和SpatialDE对SGVs的分析结果难以辨识,而利用SpaGCN方法呈现的结果更好。第五个数据集关于小鼠大脑,数据分布密集。最后的MERFISH数据集,测得基因数较少,细胞数较多。从不同的方法对六个数据集的分析结果中可以看出,SpaGCN在聚类上比stLearn、BayesSpace表现更好,且选取的SVGs比SpatialDE、SPARK方法更具有代表性。
最后李向杰从时耗、内存占用的角度对SpaGCN、SpatialDE、SPARK三种方法进行比较,SpaGCN也具有明显的优势。
最后,在提问交流环节,在线师生积极参与讨论。李向杰对研究所采取的图卷积网络模型进行了说明,并且分享了改进该模型的可行方法。

